在收集数据的时候,一般会去GEO查找,但是GEO关键字搜索一个个点非常麻烦;
于是想快速筛选结果;
刚好Biopython提供了访问NCBI Entrez的包;拼接下就写个循环就可以使用;
主要是进行字符串的提取
原文:
https://biopython-cn.readthedocs.io/zh_CN/latest/cn/chr09.html
可以访问NCBI中很多数据库,包括pubmed,gds;
所有也是可以用于文献查找的;
函数名:
** esearch **
用于搜索关键字和要选择的数据库,这里GEO使用gds;
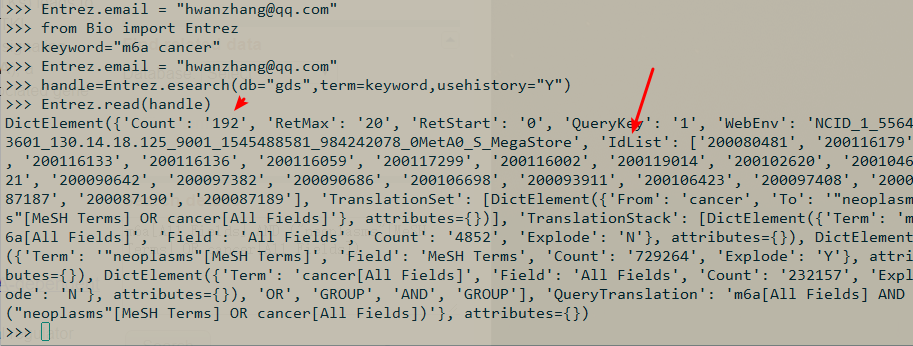
1 | handle=Entrez.esearch(db="gds",term=keyword,usehistory="Y") |
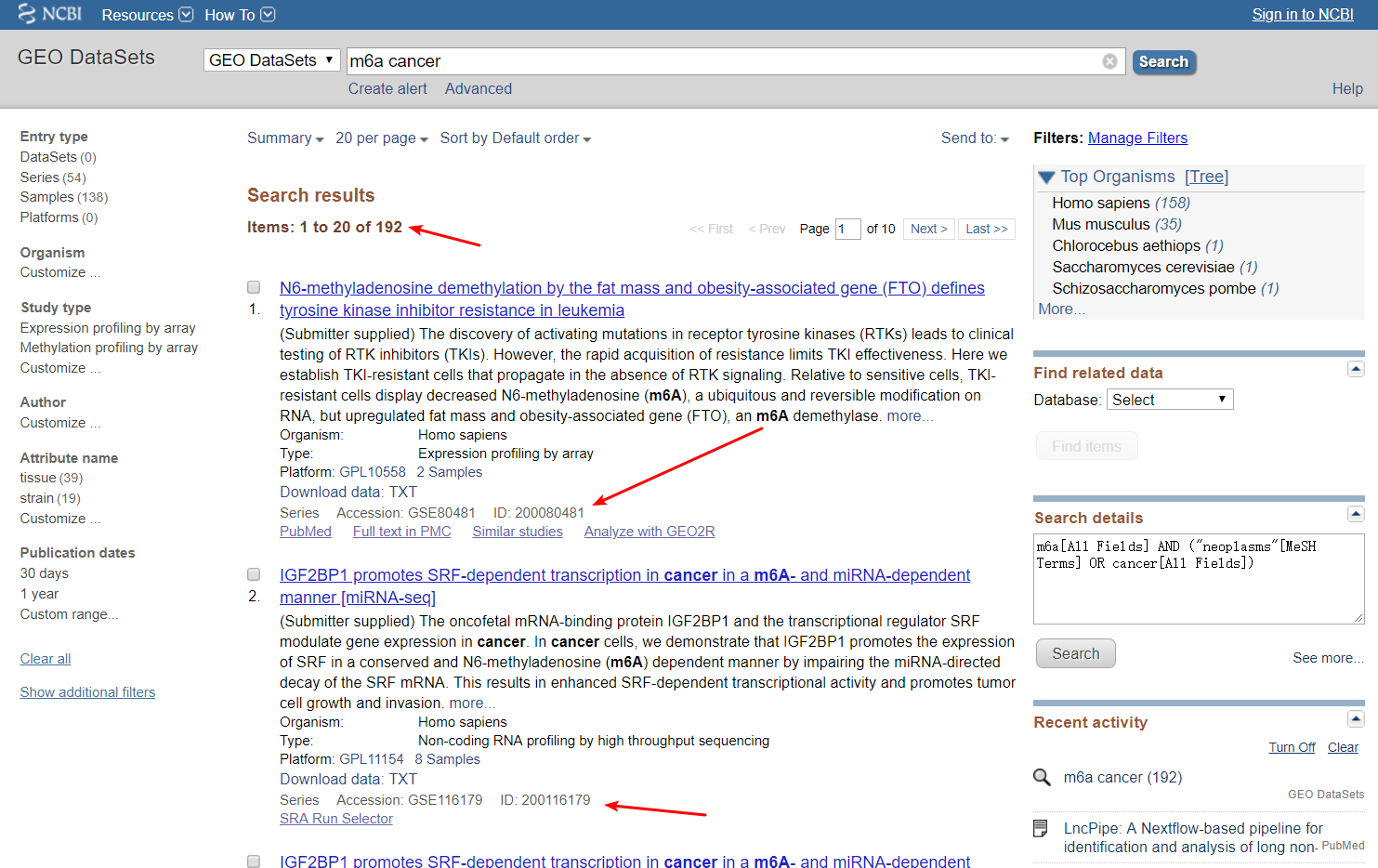
如图:GEO搜索m6A有192条信息;
Count数目对应了Items;IdList则刚好对应了ID
** esummary **
通过主要的IDs来获取摘要
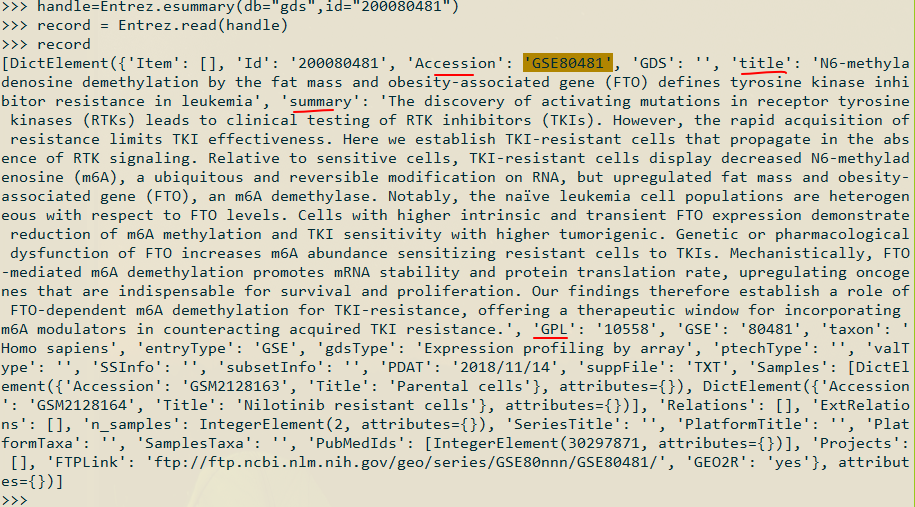
1 | handle=Entrez.esummary(db="gds",id=myid) |
使用其中第一个ID 查询信息:
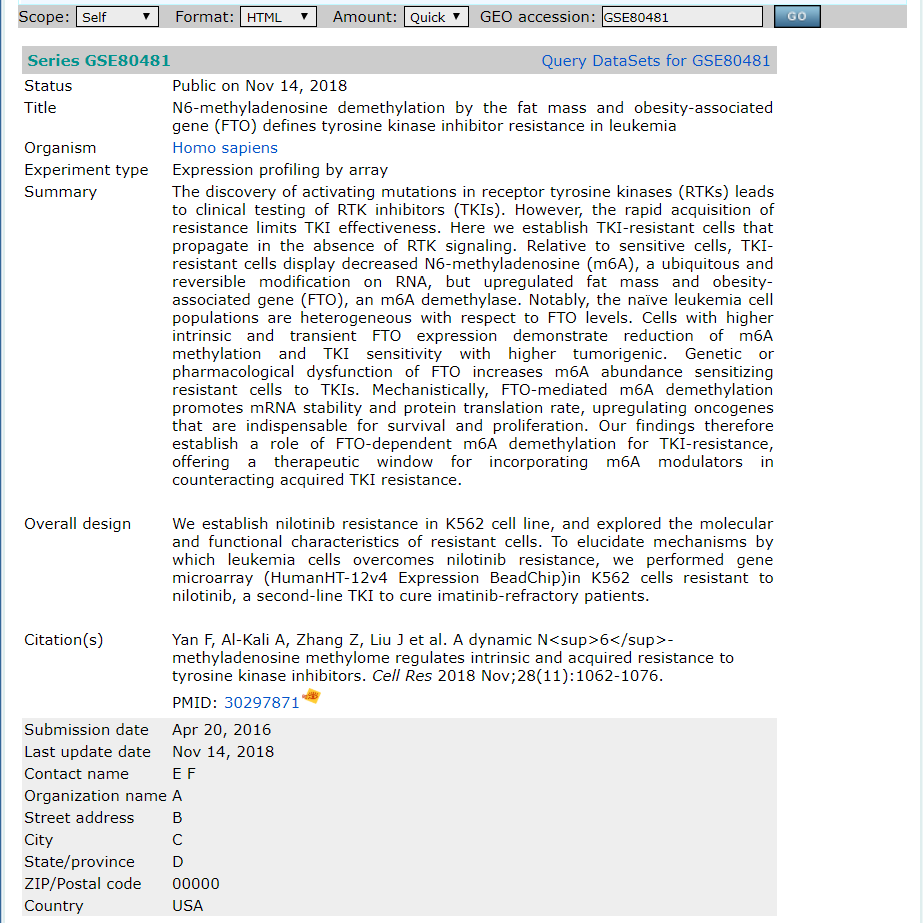
对应GEO页面搜索第一条结果GSE页面信息:
包含有GSE号,平台号,summary,以及包含的GSM号等
然后就是可以根据record进行需要的信息的提取;使用key就可以了;
当然可以继续使用GSM号查看GSM的详细信息;
之后就是拼接起来,加上循环;
但是主要问题是会断,并且每秒访问不得超过三次,之前使用多线程和异步就会出现大量超时的,明显是被拒绝访问了;容易封IP地址,所以要小心;
写好了个简单的脚步,直接运行;
两年之前做的这个,主要是为了方便自己和实验室的同学做数据收集
github 地址:
https://github.com/honvezhang/-Biopython-GEO-
使用方法:
python onlyGSE.py +关键字
比如搜索m6a cancer
1 | python onlyGSE.py m6a+cancer |